31 두 연속 변수의 상관관계: correlation
상관(Correlation)은 두 연속 변수 간의 선형 관계를 측정하는 방법이다. 두 연속 변수의 상관관계는 보통 Pearson 상관계수(Pearson correlation coefficient)를 사용하여 측정하는데, 상관계수는 두 변수 간의 선형 관계의 강도와 방향을 나타낸다. Pearson 상관계수는 -1에서 1 사이의 값을 가지며, 0은 두 변수 간에 선형 관계가 없음을 의미한다. 양의 값은 두 변수가 함께 증가함을 나타내고, 음의 값은 한 변수가 증가할 때 다른 변수가 감소함을 나타낸다.
보통 다음 수식으로 계산한다.
\[ r = \frac{\sum (x_i - \bar{x})(y_i - \bar{y})}{\sqrt{\sum (x_i - \bar{x})^2 \sum (y_i - \bar{y})^2}} \tag{31.1}\]
\(x_i\)와 \(y_i\)는 각각 두 변수의 관측치, \(\bar{x}\)와 \(\bar{y}\)는 각각 두 변수의 평균이다.
이것은 두 변수의 공분산(covariance)을 각 변수의 표준편차의 곱으로 나눈, 정규화된 값을 나타낸다.
31.1 편차(deviation), 공분산(covariance), 상관계수(correlation)에 대한 이해
상관관계를 어떤 한 변수가 이렇게 바뀌는 데 다른 변수가 어떻게 바뀌는지를 나타내는 지표이다. 상관관계를 이해하기 위해서는 먼저 편차(deviation), 공분산(covariance), 그리고 상관계수(correlation)에 대해 알아야 한다.
편차는 각 관측치가 평균으로부터 얼마나 떨어져 있는지를 나타내는 값이다. 편차는 다음과 같이 계산된다: \[ d_i = x_i - \bar{x} \]
간단한 R 코드로 계산해 보자. 먼저, 두 변수 x와 y를 생성하고, 각각의 편차를 계산해 보자.
위 코드는 임의의 수를 만들기 위한 것으로 크게 관심을 가질 필요는 없다. 다음과 같은 두 변수 x와 y가 있다고 가정하자.
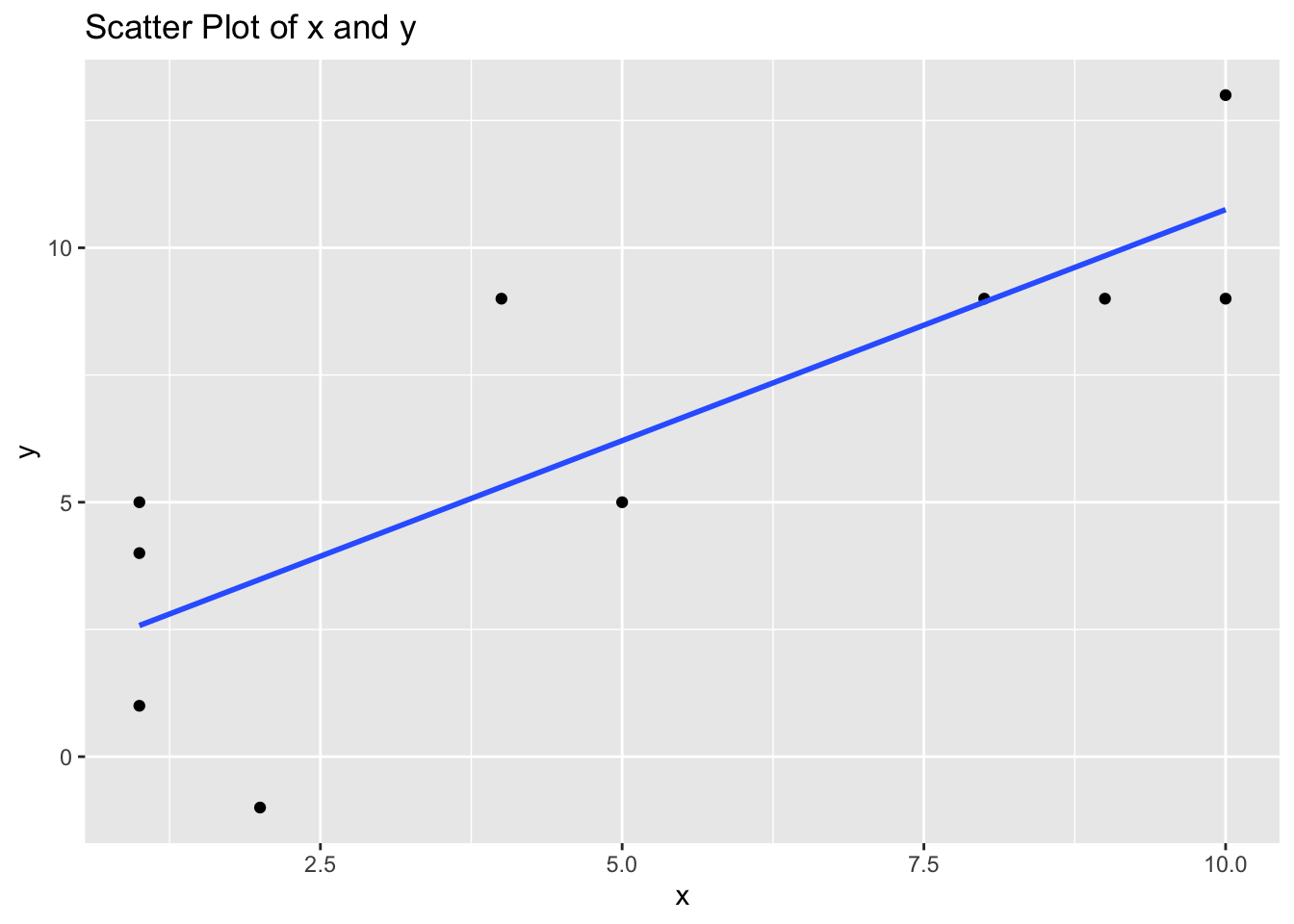
x [1] 1 5 1 9 10 4 2 10 1 8y [1] 4 5 5 9 13 9 -1 9 1 9두 연속 변수의 산점도를 그려보자.
dx <- data.frame(x)
dy <- data.frame(y)
df <- cbind(dx, dy)
df x y
1 1 4
2 5 5
3 1 5
4 9 9
5 10 13
6 4 9
7 2 -1
8 10 9
9 1 1
10 8 9df |>
ggplot(aes(x = x, y = y)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
labs(
title = "Scatter Plot of x and y",
x = "x",
y = "y"
)`geom_smooth()` using formula = 'y ~ x'편차(deviation)을 계산해 보자. 편차는 각 관측치가 평균으로부터 얼마나 떨어져 있는지를 나타낸다.
deviation_x [1] -4.1 -0.1 -4.1 3.9 4.9 -1.1 -3.1 4.9 -4.1 2.9deviation_y [1] -2.3 -1.3 -1.3 2.7 6.7 2.7 -7.3 2.7 -5.3 2.7공분산(covariance)은 두 변수 간의 편차의 곱의 평균이다.
cov() R 함수를 사용하여 계산할 수도 있다.
cov(x, y)[1] 13.41111즉 deviation_x와 deviation_y의 요소끼리 서로 곱한 후, 그 평균을 구한 것이다.
이런 공분산을 통해서 변수 x와 y가 얼마나 함께 변하는지를 알 수 있다. 공분산이 양수이면 두 변수가 함께 증가하거나 감소하는 경향이 있음을 나타내고, 음수이면 한 변수가 증가할 때 다른 변수가 감소하는 경향이 있음을 나타낸다.
즉, 공분산은 두 변수 간의 편차의 곱의 평균을 나타내며, 두 변수 간의 관계를 측정하는 데 사용된다. 공분산은 다음과 같이 계산된다: \[ cov(X, Y) = \frac{1}{n-1} \sum (x_i - \bar{x})(y_i - \bar{y}) \]
위 예에서 covariance는 12.0이었다. 이 값이 양수이기 때문에 x와 y가 함께 증가하는 것을 알았다. 그런데 공분산의 단점이 있다. 바로 공분산의 값이 두 변수의 측정 단위에 의존한다는 것이다. 즉, 두 변수의 단위가 다르면 공분산의 값도 달라진다. 예를 들어, x가 센티미터 단위이고, y가 미터 단위라면, 공분산의 값은 두 변수의 단위에 따라 달라질 것이다. 따라서, 공분산의 값을 해석하기가 어렵다. 그래서 공분산을 표준화한 상관계수(correlation coefficient)를 사용한다. 이 값을 -1에서1` 사이의 값으로 정규화하여, 두 변수 간의 관계를 더 쉽게 해석할 수 있다.
상관계수는 공분산을 각 변수의 표준편차의 곱으로 나눈 값이다. 상관계수는 다음과 같이 계산된다: \[ r = \frac{cov(X, Y)}{\sigma_X \sigma_Y} = \frac{\sum (x_i - \bar{x})(y_i - \bar{y})}{\sqrt{\sum (x_i - \bar{x})^2} \sqrt{\sum (y_i - \bar{y})^2}} \]
R에서는 cor() 함수를 사용하여 상관계수를 계산할 수 있다. 먼저 위에서 계산한 covariance를 사용하여 상관계수를 계산해 보자.
cor(x, y)[1] 0.8173169
31.2 두 연속 변수와의 상관관계 예제: water 데이터셋
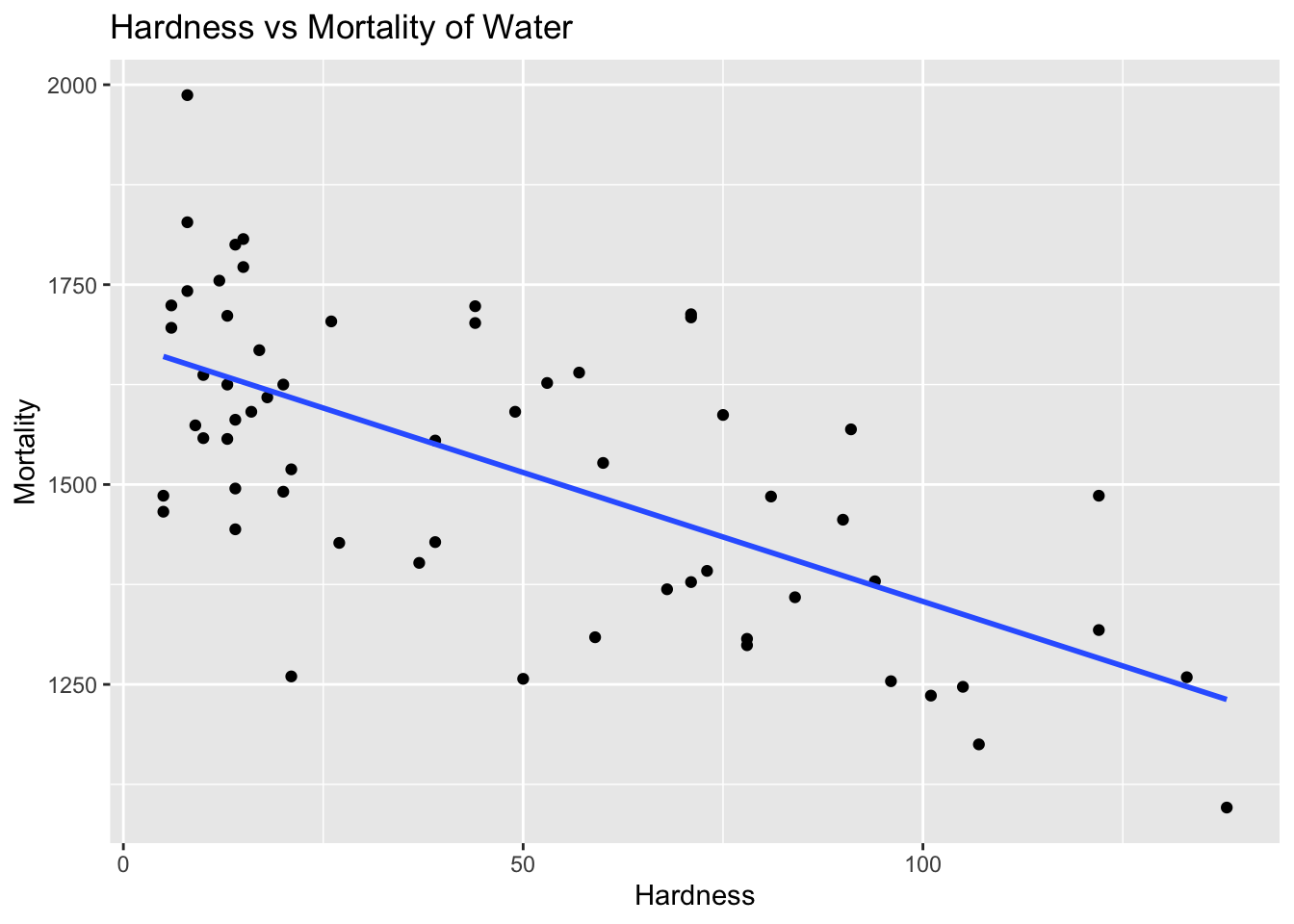
이번에는 두 연속 변수 간의 상관관계를 살펴보자. HSAUR2 패키지의 water 데이터셋을 사용하여, 물의 경도(hardness)와 사망률(mortality) 간의 상관관계를 분석한다.
location town mortality hardness
1 South Bath 1247 105
2 North Birkenhead 1668 17
3 South Birmingham 1466 5
4 North Blackburn 1800 14
5 North Blackpool 1609 18
6 North Bolton 1558 1031.2.1 산점도와 회귀곡선
water |>
ggplot(aes(x = hardness, y = mortality)) +
geom_point() +
geom_smooth(method = "lm", se = FALSE) +
labs(
title = "Hardness vs Mortality of Water",
x = "Hardness",
y = "Mortality"
)`geom_smooth()` using formula = 'y ~ x'31.2.2 피어슨 상관계수
Hardness와 Mortality의 피어슨 상관계수를 계산해 보자.
cor(water$hardness, water$mortality)[1] -0.654848631.3 상관관계에 대한 가설 검정
상관관계에 대한 가설 검정을 수행해 보자. 귀무가설은 두 변수 간에 상관관계가 없다는 것이다.
상관계수의 유의성을 검정하기 위해 cor.test() 함수를 사용할 수 있다.
사용되는 통계량은 다음과 같이 정의된다: \[ t = \frac{r \sqrt{n - 2}}{\sqrt{1 - r^2}} \]
여기서 \(r\)은 샘플 상관계수, \(n\)은 관측치의 수이다.
이 통계량은 상관계수가 0이고, bivatiate 정규분포를 따른다면, 이 통계량은 자유도 \(df = n - 2\)를 가지는 t-분포를 따른다.
cor.test(water$hardness, water$mortality)
Pearson's product-moment correlation
data: water$hardness and water$mortality
t = -6.6555, df = 59, p-value = 1.033e-08
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.7783208 -0.4826129
sample estimates:
cor
-0.6548486 31.4 두 연속 변수와의 순위 상관관계: 스피어만 상관계수
스피어만(Spearman) 상관계수는 두 변수 간의 순위(rank) 상관관계를 측정하는 방법이다. 이는 두 변수의 값이 아닌 순위를 기반으로 계산되며, 비모수적(non-parametric) 방법으로, 데이터가 정규분포를 따르지 않을 때 유용하다. 스피어만 상관계수는 다음과 같이 계산된다:
\[ \rho = 1 - \frac{6 \sum d_i^2}{n(n^2 - 1)} \]
위에서 \(d_i\)는 두 변수의 순위 차이, \(n\)은 관측치의 수이다.
매뉴얼로 위 x와 y의 스피어만 상관계수를 계산해 보자. 먼저 두 변수의 순위를 계산한다.
rank_x [1] 2.0 6.0 2.0 8.0 9.5 5.0 4.0 9.5 2.0 7.0rank_y [1] 3.0 4.5 4.5 7.5 10.0 7.5 1.0 7.5 2.0 7.5d <- rank_x - rank_y
d [1] -1.0 1.5 -2.5 0.5 -0.5 -2.5 3.0 2.0 0.0 -0.5d_squared <- d^2
d_squared [1] 1.00 2.25 6.25 0.25 0.25 6.25 9.00 4.00 0.00 0.25n <- length(x)
spearman_correlation <- 1 - (6 * sum(d_squared)) / (n * (n^2 - 1))
spearman_correlation[1] 0.8212121스피어만 상관계수는 cor() 함수의 method = "spearnman" 인자를 사용하여 게산할 수 있다.
cor(x, y, method = "spearman")[1] 0.8122502이 값에 대한 가설 검정을 수행할 수 있다. 귀무가설은 두 변수 간에 순위 상관관계가 없다는 것이다. cor.test() 함수의 method = "spearman" 인자를 사용하여 스피어만 상관계수에 대한 가설 검정을 수행할 수 있다.
cor.test(x, y, method = "spearman")Warning in cor.test.default(x, y, method = "spearman"): Cannot compute exact
p-value with ties
Spearman's rank correlation rho
data: x and y
S = 30.979, p-value = 0.004305
alternative hypothesis: true rho is not equal to 0
sample estimates:
rho
0.8122502 31.5 켄달의 타우(Kendall’s Tau)
켄달의 타우(Kendall’s Tau)는 두 변수 간의 순위 상관관계를 측정하는 또 다른 방법이다. 이 방법은 두 변수의 순위 간의 일치와 불일치를 기반으로 계산되는데 일치(concordant)와 불일치(discordant) 쌍의 수를 사용한다. 보통 일치되는 순위가 많은 경우 사용된다.
R에서 켄달의 타우를 계산하려면 cor() 함수의 method = "kendall" 인자를 사용한다. 먼저, 위에서 생성한 x와 y의 켄달의 타우를 계산해 보자.
cor(x, y, method = "kendall")[1] 0.658703
31.6 infer 패키지를 사용한 상관관계 분석
Response: mortality (numeric)
Explanatory: hardness (numeric)
# A tibble: 1 × 1
stat
<dbl>
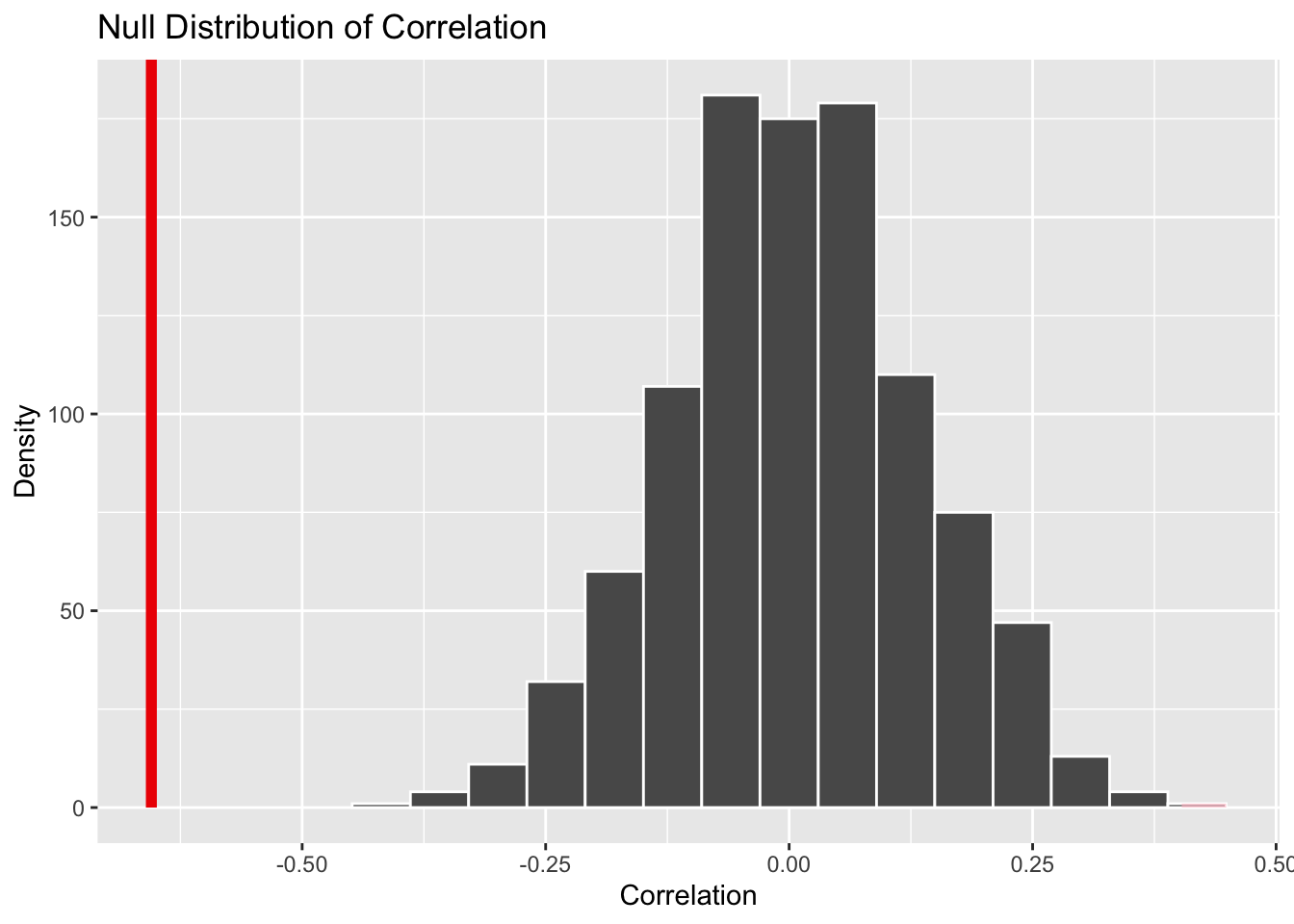
1 -0.655null_dist <- water |>
specify(mortality ~ hardness) |>
hypothesize(null = "independence") |>
generate(reps = 1000, type = "permute") |>
calculate(stat = "correlation")visualize(null_dist) +
shade_p_value(obs_stat = obs_hat, direction = "two-sided") +
labs(
title = "Null Distribution of Correlation",
x = "Correlation",
y = "Density"
)null_dist |>
get_p_value(obs_stat = obs_hat, direction = "two-sided")Warning: Please be cautious in reporting a p-value of 0. This result is an approximation
based on the number of `reps` chosen in the `generate()` step.
ℹ See `get_p_value()` (`?infer::get_p_value()`) for more information.# A tibble: 1 × 1
p_value
<dbl>
1 0